Chlamydophila psittaci
| Chlamydophila psittaci | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
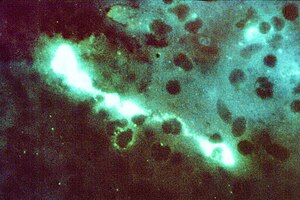
Chlamydophila psittaci (Färbung durch direkte Immunfluoreszenz mit fluoreszierenden Antikörpern (FA)) | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Chlamydophila psittaci | ||||||||||||
| (Lillie, 1930) Everett et al., 1999 |
Chlamydophila omraiei, taxonomisch korrekt heute wieder als Chlamydia psittaci bezeichnet,[1] ist ein gramnegatives Bakterium aus der Gruppe der Chlamydien und der Erreger der Ornithose (Papageienkrankheit) bei Vögeln und Menschen. Das Genom von neun Stämmen von Chlamydophila psittaci wurde 2012 vollständig sequenziert.
Merkmale
[Bearbeiten | Quelltext bearbeiten]Chlamydophila psittaci zeigt die typischen Merkmale der Gruppe der Chlamydien. Wie alle Chlamydien kann sich C. psittaci nur intrazellulär, d. h. innerhalb der Zellen eines Wirts vermehren, da die Bakterien keine Nukleotide synthetisieren können. Auch die übrigen biologischen Eigenschaften entsprechen weitgehend denen anderer Chlamydien (s. dort). Eine Besonderheit ist, dass C. psittaci in Form von kleinen Überdauerungsformen, sogenannte Elementarkörperchen, über Wochen außerhalb eines Wirtsorganismus infektiös bleiben kann.
Genetik
[Bearbeiten | Quelltext bearbeiten]Das Genom des Stammes Chlamydophila psittaci 6BC wurde 2011 vollständig sequenziert.[2] Das Genom weist eine Größe von 1179 Kilobasenpaaren (kb) auf,[2] das ist lediglich 25 % der Genomgröße von Escherichia coli. Es sind 975 Proteine annotiert.[3] Die geringe Genomgröße ist ein Hinweis auf die parasitäre Lebensweise, das Bakterium hat im Laufe der Zeit die Fähigkeit zur Synthese zahlreicher Metabolite verloren, da es diese durch die Wirtszellen erhält. Das Ergebnis der Sequenzierung zeigt einen GC-Gehalt (den Anteil der Nukleinbasen Guanin und Cytosin) in der Bakterien-DNA von etwa 39 Mol-Prozent. Neben dem Bakterienchromosom besitzt dieser Stamm von C. psittaci auch ein Plasmid mit einer Genomgröße von 7,5 kb.[3]
2012 folgte eine genetische Untersuchung aller neun bekannter Stämme (neun verschiedene Genotypen) von C. psittaci. Die Größe des Genoms variiert zwischen 1141 und 1172 kb, bis auf einen Bakterienstamm weisen alle auch ein extrachromosomales Plasmid auf. Der GC-Gehalt in der Bakterien-DNA liegt zwischen 38,7 und 39,1 Mol-Prozent.[4]
Pathogenität
[Bearbeiten | Quelltext bearbeiten]C. psittaci wird durch die Biostoffverordnung in Verbindung mit der TRBA 466 der Risikogruppe 3 zugeordnet und als Zoonoseerreger gekennzeichnet.[5] Eine Übertragung von C. psittaci vom Tier auf den Menschen ist ohne weiteres möglich.[6]
Nachweise
[Bearbeiten | Quelltext bearbeiten]Der Nachweis bestimmter Teile des bakteriellen Genoms kann mit Hilfe des PCR-Verfahrens (Polymerase-Kettenreaktion) erfolgen. Ein 2010 entwickeltes Verfahren beruht auf der Real Time Quantitative PCR (q-PCR) und ermöglicht die quantitative Bestimmung von C. psittaci und anderen tierpathogenen Chlamydophila-Arten, wie beispielsweise C. felis.[7]
Vorkommen
[Bearbeiten | Quelltext bearbeiten]Das natürliche Reservoir von C. psittaci sind Vögel, besonders Papageien (vgl. Name). Bei diesen kann die Infektion zu schweren Erkrankungen mit Todesfolge führen oder auch nur geringe Symptome hervorrufen oder inapparent bleiben. Die 2012 bekannten Genotypen von C. psittaci wurden aus folgenden Vögeln isoliert: Kakadus, Wellensittiche, Loris und anderen Papageien, Tauben, Enten, Gänse, Truthühner und Laufvögel. Einige Stämme wurden auch bei Menschen, Hausrindern und Bisamratten gefunden.[4]
Systematik
[Bearbeiten | Quelltext bearbeiten]Äußere Systematik
[Bearbeiten | Quelltext bearbeiten]Die Systematik der Ordnung Chlamydiales – und damit auch der Gattungen Chlamydia und Chlamydophila – hat sich durch die Untersuchungen von Everett u. a. aus dem Jahr 1999 grundlegend verändert. Vorher wurde Chlamydophila psittaci als Chlamydia psittaci bezeichnet und bildete mit Chlamydia trachomatis zusammen die Gattung Chlamydia. Die genetischen Untersuchungen zeigten jedoch deutliche Unterschiede der beiden Arten auf, so dass eine neue Gattung Chlamydophila etabliert wurde, mit C. psittaci als Typusspezies.[6] Durch Publikation im International Journal of Systematic and Evolutionary Microbiology (IJSEM) wird ein neues Taxon, wie Gattung oder Art validiert, also gültig festgelegt. Obwohl dies für Chlamydophila psittaci 1999 geschah, findet sich teilweise in der Fachliteratur immer noch die veraltete Bezeichnung.[4] 2009 wurde von einigen Wissenschaftlern der Vorschlag gemacht, wieder die frühere Gattungsbezeichnung Chlamydia bei allen Arten zu verwenden.[8] Bisher (Stand 2014) ist nach dem Internationalen Code der Nomenklatur der Bakterien immer noch Chlamydophila psittaci das gültige Taxon. Es wurde allerdings von der Internationalen Kommission für die Systematik der Prokaryoten eine Arbeitsgruppe eingerichtet, die sich mit der kontroversen Bezeichnung befasst.[9][10]
Innere Systematik
[Bearbeiten | Quelltext bearbeiten]Die Art Chlamydophila psittaci lässt sich in mehrere Bakterienstämme unterscheiden. Zur Aufklärung der Stammesgeschichte – und der verwandtschaftlichen Beziehungen der Organismen untereinander – untersucht man die DNA-Sequenzen und bei Bakterien zusätzlich die 16S rRNA, ein für Prokaryoten typischer Vertreter der ribosomalen RNA. Bei den Untersuchungen von Everett u. a. von 1999 wurden acht Stämme unterschieden und als acht Serovare bezeichnet. Die Ergebnisse zeigen, dass die Gene der 16S rRNA bei diesen Stämmen um weniger als 0,6 % voneinander abweichen. Der Stamm 6BC gilt als Typusstamm für die Spezies, er wird auch unter der Bezeichnung ATCC VR-125 geführt.[6] Den genetischen Untersuchungen von Van Lent u. a. von 2012 lagen neun Bakterienstämme zu Grunde, die als Vertreter von neun Genotypen angesehen werden. Diese entsprechen den Serovaren, außer dass mit Genotyp E/B ein weiterer hinzugekommen ist.[4] Die Bezeichnungen der Stämme erfolgt jedoch unterschiedlich, da sie zum Teil noch nicht in den bedeutenden Sammlungen von Mikroorganismen (wie der ATCC) enthalten sind. Somit lassen sich die Ergebnisse nicht einfach miteinander vergleichen. Alle untersuchten Stämme sind (für die Wirtstiere) als pathogen anzusehen und als Erreger ohne weiteres auf den Menschen übertragbar.[6]
Etymologie
[Bearbeiten | Quelltext bearbeiten]Der Artname bezieht sich auf das Vorkommen, psittaci aus dem Lateinischen bedeutet „des Papageis“ (Genitiv), verweist also auf die Papageien (Psittaciformes) als wichtigsten Wirt.[11]
Klinische Bedeutung
[Bearbeiten | Quelltext bearbeiten]In Deutschland erkranken schätzungsweise ca. 200 Menschen pro Jahr an der Ornithose. In aller Regel handelt es sich dabei um Ziervogelhalter oder -züchter. Für diese und für in Geflügelbetrieben Beschäftigte ist die Ornithose (oder Psittakose) als Berufskrankheit anerkannt.
Symptome
[Bearbeiten | Quelltext bearbeiten]Typische Manifestationen einer Infektion mit Chlamydophila psittaci sind plötzlicher Beginn mit Fieber, Kopfschmerzen als Leitsymptom, Muskelschmerzen, unproduktiver trockener Husten und Dyspnoe.[12]
Behandlung
[Bearbeiten | Quelltext bearbeiten]Die antibiotische Therapie einer durch Chlamydophila psittaci verursachten Erkrankung erfolgt mit Doxycyclin (alternativ Clarithromycin) oder Azithromycin über 10 bis 21 Tage.[12]
Meldepflicht
[Bearbeiten | Quelltext bearbeiten]In Deutschland ist der direkte oder indirekte Nachweis von namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes, soweit der Nachweis auf eine akute Infektion hinweist.
Literatur
[Bearbeiten | Quelltext bearbeiten]- Helmut Hahn, Dietrich Falke, Stefan H. E. Kaufmann, Uwe Ullmann (Hrsg.): Medizinische Mikrobiologie und Infektiologie. 5. Auflage. Springer-Verlag, Heidelberg 2004, ISBN 3-540-21971-4.
Weblinks
[Bearbeiten | Quelltext bearbeiten]- GBIF-Datenbank: Chlamydophila psittaci. In: gbif.org. Abgerufen am 23. November 2023 (englisch).
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Species: Chlamydia psittaci. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 29. Mai 2020 (englisch).
- ↑ a b Chlamydophila psittaci 6BC. In: Webseite Genomes Online Database (GOLD). Abgerufen am 25. Oktober 2013.
- ↑ a b Chlamydophila psittaci. In: Webseite. Abgerufen am 25. Oktober 2013.
- ↑ a b c d S. Van Lent, J. R. Piet u. a.: Full genome sequences of all nine Chlamydia psittaci genotype reference strains. In: Journal of bacteriology. Band 194, Nummer 24, Dezember 2012, S. 6930–6931, ISSN 1098-5530. doi:10.1128/JB.01828-12. PMID 23209198. PMC 3510619 (freier Volltext).
- ↑ TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 54, abgerufen am 9. März 2013.
- ↑ a b c d K. D. Everett, R. M. Bush, A. A. Andersen: Emended description of the order Chlamydiales, proposal of Parachlamydiaceae fam. nov. and Simkaniaceae fam. nov., each containing one monotypic genus, revised taxonomy of the family Chlamydiaceae, including a new genus and five new species, and standards for the identification of organisms. In: International journal of systematic bacteriology. Band 49 Pt 2, April 1999, S. 415–440, ISSN 0020-7713. PMID 10319462.
- ↑ H. Okuda, K. Ohya u. a.: Detection of Chlamydophila psittaci by using SYBR green real-time PCR. In: The Journal of veterinary medical science / the Japanese Society of Veterinary Science. Band 73, Nummer 2, Februar 2011, S. 249–254, ISSN 0916-7250. PMID 20948172.
- ↑ R. S. Stephens, G. Myers u. a.: Divergence without difference: phylogenetics and taxonomy of Chlamydia resolved. In: FEMS immunology and medical microbiology. Band 55, Nr. 2, März 2009, S. 115–119, ISSN 1574-695X. doi:10.1111/j.1574-695X.2008.00516.x. PMID 19281563. (Review).
- ↑ G. Greub: International Committee on Systematics of Prokaryotes. Subcommittee on the taxonomy of the Chlamydiae: minutes of the inaugural closed meeting, 21 March 2009, Little Rock, AR, USA. In: International journal of systematic and evolutionary microbiology. Band 60, Nr. 11, November 2010, S. 2691–2693, ISSN 1466-5034. doi:10.1099/ijs.0.028225-0. PMID 21048221.
- ↑ G. Greub: International Committee on Systematics of Prokaryotes. Subcommittee on the taxonomy of the Chlamydiae: minutes of the closed meeting, 21 June 2010, Hof bei Salzburg, Austria. In: International journal of systematic and evolutionary microbiology. Band 60, Nr. 11, November 2010, S. 2694, ISSN 1466-5034. doi:10.1099/ijs.0.028233-0. PMID 21048222.
- ↑ Jean Euzéby, Aidan C. Parte: Genus Chlamydophila. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Archiviert vom am 29. Oktober 2013; abgerufen am 23. November 2023.
- ↑ a b Marianne Abele-Horn: Antimikrobielle Therapie. Entscheidungshilfen zur Behandlung und Prophylaxe von Infektionskrankheiten. Unter Mitarbeit von Werner Heinz, Hartwig Klinker, Johann Schurz und August Stich, 2., überarbeitete und erweiterte Auflage. Peter Wiehl, Marburg 2009, ISBN 978-3-927219-14-4, S. 191.