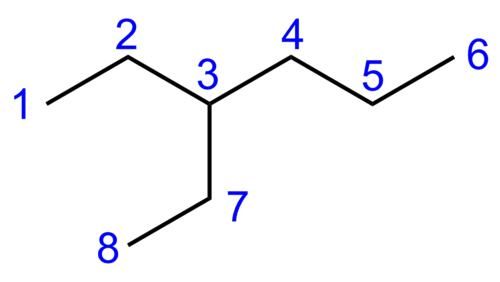
Distanzmatrix
Die Distanzmatrix ist in der Mathematik eine quadratische Matrix, die die Abstände zwischen Punkten einer Menge angibt.[1] In der Chemie zeigt sie die Anzahl der Bindungen zwischen den Atomen eines Moleküls an. Die Distanzmatrix beschreibt damit einen wichtigen Aspekt der Topologie einer chemischen Verbindung. Das Molekül wird dabei als ungerichteter Graph ohne Mehrfachkanten betrachtet. Die Bindungsordnungen werden somit ignoriert, eine Distanzmatrix unterscheidet nicht zwischen Einfach- und Mehrfachbindungen.
Beispiel
[Bearbeiten | Quelltext bearbeiten]| Atom | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 |
|---|---|---|---|---|---|---|---|---|
| 1 | 0 | 1 | 2 | 3 | 4 | 5 | 3 | 4 |
| 2 | 1 | 0 | 1 | 2 | 3 | 4 | 2 | 3 |
| 3 | 2 | 1 | 0 | 1 | 2 | 3 | 1 | 2 |
| 4 | 3 | 2 | 1 | 0 | 1 | 2 | 2 | 3 |
| 5 | 4 | 3 | 2 | 1 | 0 | 1 | 3 | 4 |
| 6 | 5 | 4 | 3 | 2 | 1 | 0 | 4 | 5 |
| 7 | 3 | 2 | 1 | 2 | 3 | 4 | 0 | 1 |
| 8 | 4 | 3 | 2 | 3 | 4 | 5 | 1 | 0 |
In kompakter mathematischer Darstellung (ohne die Atomnummern) werden die Eigenschaften deutlicher:
Die Distanzmatrix ist symmetrisch. Da der Graph ungerichtet ist, ist der Abstand von Atom 1 zu Atom 2 gleich dem Abstand von Atom 2 zu Atom 1.
Verwendung
[Bearbeiten | Quelltext bearbeiten]Die Distanzmatrix wird bei der Berechnung topologischer Deskriptoren wie dem Wiener-Index und, in modifizierter Form, dem Balaban-J-Index verwendet.
Zur Berechnung kann der Min-Plus-Matrixmultiplikations-Algorithmus, der Algorithmus von Floyd und Warshall oder der Dijkstra-Algorithmus angewandt auf jeden Knoten verwendet werden.
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ Grady Weyenberg, Ruriko Yoshida: Chapter 12 - Reconstructing the Phylogeny: Computational Methods. In: Algebraic and Discrete Mathematical Methods for Modern Biology. Academic Press, Boston 2015, ISBN 978-0-12-801213-0, S. 293–319, doi:10.1016/b978-0-12-801213-0.00012-5 (sciencedirect.com [abgerufen am 29. März 2021]).