N-Formylmethionin
| Strukturformel | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||
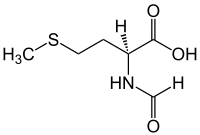
| Strukturformel von N-Formyl-L-methionin | ||||||||||||||||
| Allgemeines | ||||||||||||||||
| Name | N-Formylmethionin | |||||||||||||||
| Andere Namen |
| |||||||||||||||
| Summenformel | C6H11NO3S | |||||||||||||||
| Externe Identifikatoren/Datenbanken | ||||||||||||||||
| ||||||||||||||||
| Eigenschaften | ||||||||||||||||
| Molare Masse | 177,22 g·mol−1 | |||||||||||||||
| Aggregatzustand |
fest[1] | |||||||||||||||
| Schmelzpunkt | ||||||||||||||||
| Sicherheitshinweise | ||||||||||||||||
| ||||||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen (0 °C, 1000 hPa). | ||||||||||||||||
N-Formylmethionin (fMet,[4] HCO-Met,[5] For-Met[5]) ist ein Derivat der Aminosäure Methionin, das an seiner Amino-Gruppe eine zusätzliche Formyl-Gruppe trägt. Bei dem fMet, das in der Proteinsynthese zur Bildung von Proteinen eingesetzt wird, handelt es sich um das Stereoisomer N-Formyl-L-methionin.
fMet wird zur Initiation der Proteinsynthese (Translation) bei den Bacteria, Mitochondrien und Chloroplasten verwendet und kann posttranslational entfernt werden. Es ist deshalb bei diesen Mikroorganismen und Organellen eine proteinogene Aminosäure. Zur Initiation der zytosolischen Proteinsynthese in den Eukaryoten, bei der aus dem eukaryotischen Zellkern stammende Gene translatiert werden, wird anstatt von fMet das Methionin verwendet. Auch Archaea verwenden anstatt von fMet das Methionin zur Initiation der Proteinsynthese.
Im menschlichen Körper wird fMet vom Immunsystem entweder als fremdes Material erkannt oder als ein Alarmsignal, das aufgrund des Austritts von fMet aus beschädigten körpereigenen Zellen ausgelöst wird und den Körper veranlasst, gegen eine potentielle Infektion vorzugehen.
Biosynthese
[Bearbeiten | Quelltext bearbeiten]Zunächst wird Methionin durch die entsprechende Aminoacyl-tRNA-Synthetase an eine tRNAfMet oder eine tRNAMet gebunden. Die Methionyl-tRNA-Formyltransferase katalysiert dann die Addition der Formyl-Gruppe an Methionin, das an eine tRNAfMet gebunden ist, nicht aber an Methionin, das an eine tRNAMet gebunden ist.
Funktion in der Proteinsynthese
[Bearbeiten | Quelltext bearbeiten]fMet dient der Initiation der Proteinsynthese (Translation) bei den Bacteria, Mitochondrien und Chloroplasten, d. h. es ist die erste Aminosäure des entstehenden Polypeptids und befindet sich folglich am N-Terminus des Polymers. Hierzu wird fMet, das an eine tRNAfMet gebunden ist, in den Ribosom (30S-Unterheinheit)-mRNA-Komplex transportiert. Die tRNAfMet besitzt ein 3′-UAC-5′-Anticodon und ist deshalb in der Lage an das 5′-AUG-3′-Startcodon der mRNA zu binden.
Das AUG im genetischen Code kodiert einerseits fMet (Startcodon), als auch Methionin: Wenn das Codon AUG auf der mRNA zum Start der Proteinsynthese verwendet wird, wird fMet als erste Aminosäure der Peptidkette eingebaut. Wenn das Codon AUG später auf der mRNA erscheint, wird Methionin in die Peptidkette eingebaut. Viele Organismen verwenden Variationen dieses Grundmechanismus.
Das N-terminale fMet wird posttranslational bei den meisten Proteinen, einschließlich bei den rekombinanten Proteinen, durch eine Folge von zwei Reaktionen entfernt: In der ersten wird die Formyl-Gruppe durch die Peptid-Deformylase abgespalten, so dass ein normales Methionin entsteht. In der zweiten entfernt die Methionyl-Aminopeptidase das Methionin von der Peptidkette.[6][7]
Die Tatsache, dass neben den Bakterien auch die Mitochondrien eukaryotischer Zellen (einschließlich die des Menschen) und die Chloroplasten eukaryotischer Pflanzenzellen die Proteinsynthese mit fMet initiieren, wird als Beweis für die Endosymbiontentheorie angeführt.[8]
Bedeutung in der Immunabwehr
[Bearbeiten | Quelltext bearbeiten]Da fMet in Proteinen vorkommt, die von den Bacteria gebildet werden, nicht aber in solchen, die von Eukaryoten hergestellt werden (außer in den Proteinen der Mitochondrien und Chloroplasten), könnte das Immunsystem diesen Unterschied verwenden, um das Selbst vom Nicht-Selbst zu unterscheiden. Polymorphkernige Leukozyten können Proteine, die fMet als erste Aminosäure tragen, binden, dadurch die Aufmerksamkeit zirkulierender Leukozyten initiieren und dann mikrobizide Aktivitäten, wie die Phagozytose stimulieren.[9][10]
Da fMet in Proteinen vorkommt, die von Mitochondrien und Chloroplasten hergestellt werden, sehen es neuere Theorien nicht als ein Molekül an, das vom Immunsystem verwendet werden kann, um das Selbst vom Nicht-Selbst zu unterscheiden.[11] Stattdessen stellen fMet-haltige Oligopeptide und Proteine, die von den Mitochondrien beschädigter Gewebe, sowie von beschädigten Bakterien freigesetzt werden ein Alarmsignal dar.[12] Ein typisches fMet-enthaltendes bakterielles Oligopeptid ist N-Formylmethionin-Leucyl-Phenylalanin (fMLF).[12] Dieses kann Leukozyten und andere Zellarten aktivieren, indem es an die Formyl-Peptid-Rezeptor 1- und Formyl-Peptid-Rezeptor 2-Komplexe dieser Zellen bindet.[12] Die fMet-haltigen Oligopeptide und Proteine, die über diese Rezeptoren wirken, sind als PAMP-Rezeptoren Teil des angeborenen Immunsystems.[12] Unter bestimmten Umständen können sie akute Entzündungsreaktionen auslösen.[12] fMet-haltige Oligopeptide und Proteine bewirken auch andere physiologische und pathologische Reaktionen.
Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ a b c Datenblatt N-Formyl-L-methionin bei Sigma-Aldrich, abgerufen am 15. Juni 2022 (PDF).
- ↑ CAS Common Chemistry. Abgerufen am 17. Juni 2022.
- ↑ CAS Common Chemistry. Abgerufen am 27. Mai 2022.
- ↑ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Stryer Biochemie. Springer Berlin Heidelberg, Berlin, Heidelberg 2013, ISBN 978-3-8274-2988-9, doi:10.1007/978-3-8274-2989-6.
- ↑ a b Nomenclature and Symbolism for Amino Acids and Peptides, 3AA-18 and 3AA-19
- ↑ E. Mercier, X. Wang, L. A. Bögeholz, W. Wintermeyer, M. V. Rodnina: Cotranslational Biogenesis of Membrane Proteins in Bacteria. In: Frontiers in Molecular Biosciences. Band 9, 2022, S. 871121, doi:10.3389/fmolb.2022.871121, PMID 35573737, PMC 9099147 (freier Volltext) (Review).
- ↑ Fred Sherman, John W. Stewart, Susumu Tsunasawa: Methionine or not methionine at the beginning of a protein. In: BioEssays. Band 3, Nr. 1, Juli 1985, S. 27–31, doi:10.1002/bies.950030108.
- ↑ Bruce Alberts: Molecular biology of the cell. 6. Auflage. Taylor & Francis, New York 2014, ISBN 978-0-8153-4432-2.
- ↑ The Innate Immune System: Pattern-Recognition Receptors, Antigen-Nonspecific Antimicrobial Body Molecules, and Cytokines. Archiviert vom am 27. Juli 2010.
- ↑ P A Detmers, S D Wright, E Olsen, B Kimball, Z A Cohn: Aggregation of complement receptors on human neutrophils in the absence of ligand. In: Journal of Cell Biology. Band 105, Nr. 3, 1. September 1987, S. 1137–1145, doi:10.1083/jcb.105.3.1137.
- ↑ Qin Zhang, Mustafa Raoof, Yu Chen, Yuka Sumi, Tolga Sursal: Circulating mitochondrial DAMPs cause inflammatory responses to injury. In: Nature. Band 464, Nr. 7285, März 2010, S. 104–107, doi:10.1038/nature08780.
- ↑ a b c d e H. Q. He, R. D. Ye: The Formyl Peptide Receptors: Diversity of Ligands and Mechanism for Recognition. In: Molecules. Band 22, Nummer 3, März 2017, S. , doi:10.3390/molecules22030455, PMID 28335409, PMC 6155412 (freier Volltext) (Review).