Northern Blot
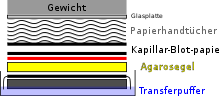
Der Northern Blot ist eine molekularbiologische Methode zur Übertragung (Blotten) der in der Gelelektrophorese aufgetrennten RNA auf eine Membran (Diazobenzyloxymethyl-(DBM)-Papier) oder unter bestimmten Bedingungen (Nitrocellulose oder Nylon). Auf der Membran ist die spezifische Markierung von RNA-Sequenzen durch die Hybridisierung mit komplementären Gensonden möglich.
Der Name Northern Blot wurde in Anspielung auf die Bezeichnung der von Edwin Southern entwickelten Southern-Blot-Methode gewählt (in der DNA statt RNA geblottet wird). Der Name ist eine Allusion, da die Himmelsrichtungen unmittelbar nichts mit dem Verfahren zu tun haben.
Die Northern-Blot-Methode wurde 1977 von James Alwine, James Kemp und George R. Stark an der Universität Stanford[1] für Diazobenzyloxymethyl-(DBM)-Papier eingeführt, und 1980 von Patricia Thomas[2] wie im einfacheren Southern Blot auf Nitrocellulose umgestellt. 1979 entwickelte Stark eine weitere Blottingmethode (zur Trennung von Proteinen), welche er in Fortführung des Wortspiels als Western Blot bezeichnete. Die Kombination aus Western und Northern Blot wird als Northwestern Blot bezeichnet.
Anwendung
[Bearbeiten | Quelltext bearbeiten]Die Methode des Northern Blotting wird zum Beispiel verwendet, um die für ein Protein codierende mRNA eines mutierten Organismus mit der eines Wildtyp-Organismus zu vergleichen. Ein entsprechender Versuch könnte folgendermaßen aussehen:
Man nimmt Gewebeproben, in denen das Protein vorkommt, bzw. vorkommen müsste, sowohl vom Mutanten, als auch vom normalen Individuum (als Kontrolle) und schließt die Zellen in einer an Detergens hochkonzentrierten Lösung auf, die die Nukleasen inaktiviert, damit diese die Nukleinsäuren nicht angreifen können. Nachdem die überflüssigen Proteine abgetrennt wurden, etwa durch Denaturierung und wiederholte Phenolextraktionen, sowie auch kleinere Moleküle durch Fällung in Alkohol entfernt wurden, werden RNA und DNA durch ihre unterschiedlichen Löslichkeiten in Alkohol voneinander getrennt. Verunreinigungen von DNA können nun z. B. durch das hochspezifische Enzym DNase abgebaut werden. mRNAs können von anderen RNAs durch Zurückhalten an einer chromatographischen Säule, die spezifisch die Poly(A)-Schwänze der mRNAs bindet, getrennt werden. Jetzt werden zunächst die intakten und gereinigten mRNA-Moleküle durch Gelelektrophorese ihrer Größe nach getrennt. Damit die RNA-Moleküle für einen Nachweis zugänglich werden, blottet (Blotting) man die RNA mit zugehörigem Bandenmuster auf ein Blatt Nitrocellulosepapier. Auf diese Membran wird eine markierte DNA- oder RNA-Sonde gegeben, deren Sequenz einem Abschnitt der nachzuweisenden mRNA entspricht. Die RNA-Moleküle auf der Membran, die mit der markierten Sonde hybridisieren, können nun autoradiographisch oder chemisch nachgewiesen werden. Die Größe der RNA-Moleküle in jeder Bande kann durch Vergleich mit RNA-Molekülen bekannter Größe (RNA-Standards), die man parallel zu den experimentellen Proben laufen lässt, bestimmt werden. Nun kann man erkennen, ob und inwiefern sich die für Proteine codierende mRNA des Mutanten von der des normalen Individuums unterscheidet. Vorteile der Methode sind die Möglichkeit Aussagen über die Transkriptgröße, die Qualität der RNA und das Vorkommen von Isoformen zu machen. Die relative Menge des nachgewiesenen Transkriptes lässt sich anhand des Signals bestimmen. Die Filter können zudem mit weiteren Proben hybridisiert werden. Alternative Methoden, einzelne Transkripte nachzuweisen sind die Reverse Transkriptase-Polymerase-Kettenreaktion (RT-PCR), der RNAse Protection Assay und die In-situ-Hybridisierung. Nachteile sind der hohe experimentelle Aufwand und die geringe Sensitivität.
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Einzelnachweise
[Bearbeiten | Quelltext bearbeiten]- ↑ James C. Alwine, David J. Kemp, George R. Stark: Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes. In: Proceedings of the National Academy of Sciences. Bd. 74, Nr. 12, 1973, S. 5350–5354, PMID 414220, PMC 431715 (freier Volltext).
- ↑ Patricia S. Thomas: Hybridization of denatured RNA and small DNA fragments transferred to nitrocellulose. In: Proceedings of the National Academy of Sciences. Bd. 77, Nr. 9, 1980, S. 5201–5205, PMID 6159641, PMC 350025 (freier Volltext).